
。 当RNA聚合酶全酶最初与DNA相结合时,它占据的长度 约75-80bp,从启动子的一55至十20。RNA聚合酶全酶 的长径为16urn(160A),只能盖一50bp的DNA,这 表明全酶结合的DNA必有某种程度的弯曲。在转录起始 阶段结束时,。因子被释放,RNA聚合酶形状发生改变, 失去与DNA一55至一35区域间的接触,此时核心酶覆盖的 长度约为60bp。 -50-40-30-20-101102030 电泳方向 654 -50-40-30-20-101102030 3321 NNOXOOOOXN 50-40-30-20-101102030 432 654 蛋白质结合部位 不被DNA酶水解 对照 图36-4足迹法测定DNA上蛋白质的 图36-5大肠杆菌RNA聚合酶在载录起始阶段 结合部位 缩短覆盖DNA的长度
• 当 RNA聚合酶全酶最初与 DNA相结合时,它占据的长度 约 75-80 bp,从启动子的一55至十20。RNA聚合酶全酶 的长径为 16 urn(160A),只能盖一50 bp的 DNA,这 表明全酶结合的 DNA必有某种程度的弯曲。在转录起始 阶段结束时,。因子被释放,RNA聚合酶形状发生改变, 失去与DNA-55至一35区域间的接触,此时核心酶覆盖的 长度约为 60 bp
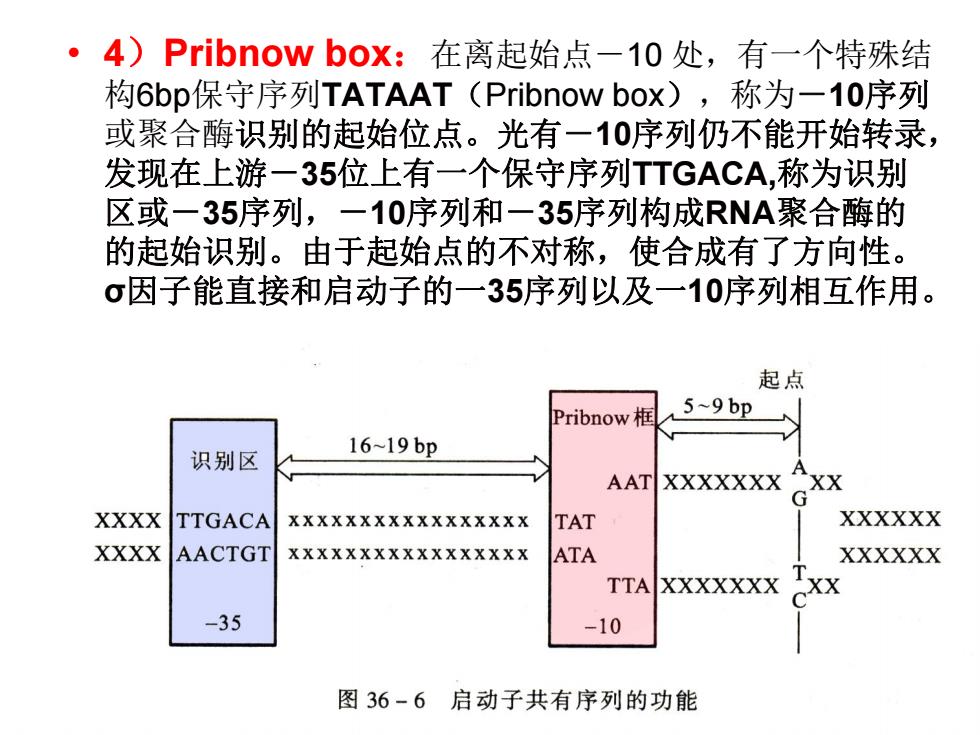
4)Pribnow box:在离起始点一10处,有一个特殊结 构6bp保守序列TATAAT(Pribnow box),称为一10序列 或聚合酶识别的起始位点。光有一10序列仍不能开始转录, 发现在上游一35位上有一个保守序列TTGACA,称为识别 区或一35序列,一10序列和一35序列构成RNA聚合酶的 的起始识别。由于起始点的不对称,使合成有了方向性。 σ因子能直接和启动子的一35序列以及一10序列相互作用。 起点 Pribnow框 5~9bp 识别区 16~19bp AAT XXXXXXX XX G XXXX TTGACA XXXXXXXXXXXXXXXXX TAT XXXXXX XXXX AACTGT XXXXXXXXXXXXXXXXX ATA XXXXXX TTA XXXXXXX -35 -10 图36-6启动子共有序列的功能
• 4)Pribnow box:在离起始点-10 处,有一个特殊结 构6bp保守序列TATAAT(Pribnow box),称为-10序列 或聚合酶识别的起始位点。光有-10序列仍不能开始转录, 发现在上游-35位上有一个保守序列TTGACA,称为识别 区或-35序列,-10序列和-35序列构成RNA聚合酶的 的起始识别。由于起始点的不对称,使合成有了方向性。 σ因子能直接和启动子的一35序列以及一10序列相互作用

4、RNA聚合酶的作用方式 ·1)以双链DNA为模板,天然双螺旋DNA为模 板要比单链DNA更有效。以全保留方式转录, 已合成的RNA离开DNA链。 转录泡,17bp 解旋 DN 重旋 RNA RNA-DNA活性中心 杂交体,8bp 酶的移动方向
• 1)以双链DNA为模板,天然双螺旋DNA为模 板要比单链DNA更有效。以全保留方式转录, 已合成的RNA离开DNA链。 4、RNA聚合酶的作用方式

5、终止子和终止因子 ·1)细菌和真核生物转录一旦起始,通常都能 继续下去,直至转录完成而终止。但在转录的 延伸阶段RNA聚合酶遇到障碍会停顿和受阻, 酶脱离模板即终止。转录结束,RNA聚合酶和 RNA转录产物即被释放。提供转录停止信号的 DNA序列称为终止子,协助RNA聚合酶识别终止 信号的辅助因子则称为终止因子p因子
• 1)细菌和真核生物转录一旦起始,通常都能 继续下去,直至转录完成而终止。但在转录的 延伸阶段RNA聚合酶遇到障碍会停顿和受阻, 酶脱离模板即终止。转录结束,RNA聚合酶和 RNA转录产物即被释放。提供转录停止信号的 DNA序列称为终止子,协助RNA聚合酶识别终止 信号的辅助因子则称为终止因子ρ因子。 5、终止子和终止因子

·DNA的转录终止信号可被RAN聚合酶所识别。 在转录过程中,RNA聚合酶沿着模板链向前移 动。所有原核生物的终止子在终止点之前均有 一个回文结构,其产生的RNA可形成由茎环构 成的发夹结构。该结构可使聚合酶减慢移动或 暂停RNA的合成
• DNA的转录终止信号可被RAN聚合酶所识别。 在转录过程中,RNA聚合酶沿着模板链向前移 动。所有原核生物的终止子在终止点之前均有 一个回文结构,其产生的RNA可形成由茎环构 成的发夹结构。该结构可使聚合酶减慢移动或 暂停RNA的合成