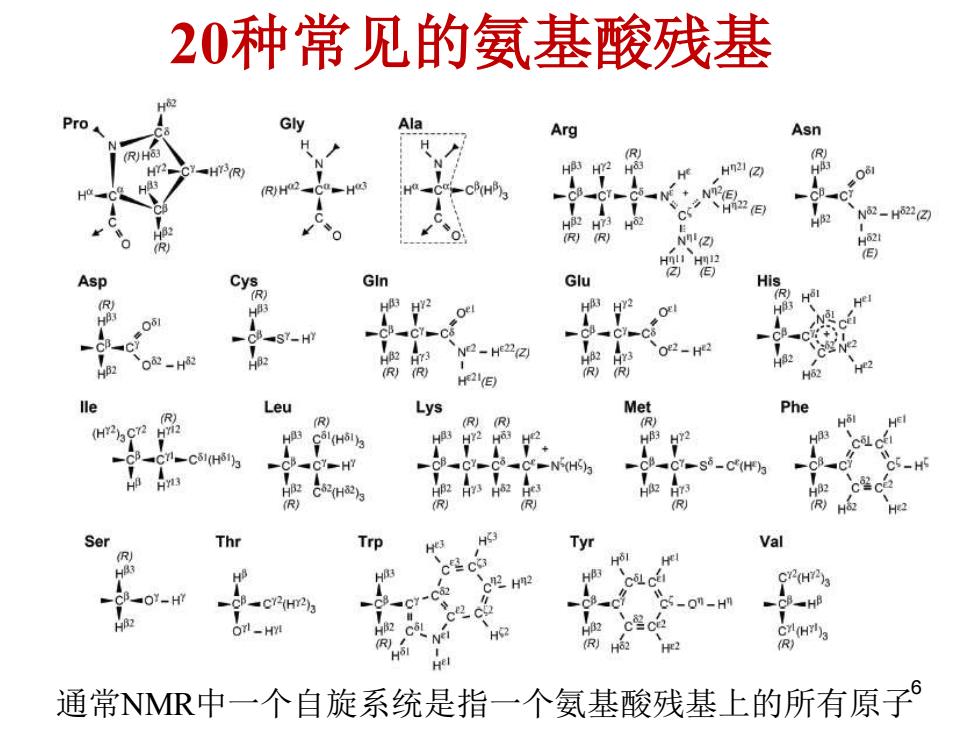
20种常见的氨基酸残基 Pro Arg Asn R Hn21 (Z) Ho (E) HB2 N2-H22☑ Asp Cys GIn Glu Hi R (R) H72 83 SY-HY 2 N2-H22Z☒ R HI(E) Ho Leu Lys Met Phe (R) (R)(R) H23C72 C(H51) HB3 -c51H313 C-NHS方 _C(H)3 B2 R R) Ser Thr Trp Val =c2H23 H C(H)3 R) 通常NMR中一个自旋系统是指一个氨基酸残基上的所有原子
通常NMR中一个自旋系统是指一个氨基酸残基上的所有原子 20种常见的氨基酸残基 6

NMR解析蛋白质溶液结构 ·测定原子(氢原子)之间的距离信息和其他约 束信息,得到空间结构模型 ·化学结构(氨基酸序列,即一级结构)己知, 测定空间结构(三级结构,四级结构) 7
NMR解析蛋白质溶液结构 • 测定原子(氢原子)之间的距离信息和其他约 束信息,得到空间结构模型 • 化学结构(氨基酸序列,即一级结构)已知, 测定空间结构(三级结构,四级结构) 7
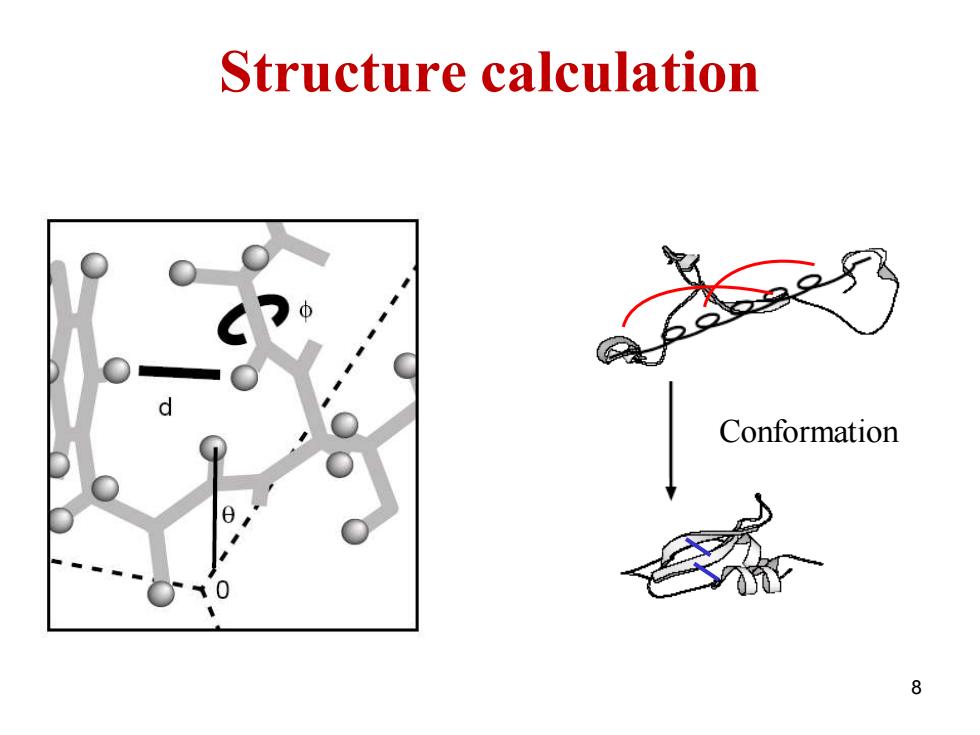
Structure calculation Conformation 8
Structure calculation Conformation 8

结构计算 ·基本方法 一由实验得到各种构象约束信息:距离,二面角 ·约束信息是不完备的 ·约束信息是不精确的 一计算满足这些约束条件的构象 。距离几何(Distance Geometry) ·约束条件下的分子动力学模拟(Restrained Molecular Dynamics Simulation) 。模拟退火(Simulated Annealing) 9
结构计算 • 基本方法 – 由实验得到各种构象约束信息:距离,二面角 • 约束信息是不完备的 • 约束信息是不精确的 – 计算满足这些约束条件的构象 • 距离几何(Distance Geometry) • 约束条件下的分子动力学模拟(Restrained Molecular Dynamics Simulation) • 模拟退火(Simulated Annealing) 9

NMR experimental observables providing structural information Backbone conformation from chemical shifts (Chemical Shift Index-CSI):W, Distance restraints from NOEs Hydrogen bond restraints Backbone and side chain dihedral angle restraints from scalar couplings Orientation restraints from residual dipolar couplings 10
10 NMR experimental observables providing structural information • Backbone conformation from chemical shifts (Chemical Shift Index - CSI): , • Distance restraints from NOEs • Hydrogen bond restraints • Backbone and side chain dihedral angle restraints from scalar couplings • Orientation restraints from residual dipolar couplings